第五章入门
模型方程可以通过文本字符串、单独的模型文件或R表达式来指定。微分方程和代数方程都是允许的。微分方程由d/dt(var_name) =指定。不同方程间可以用英文分号分隔开。
加载rxode2包并编译模型:
library(rxode2)#> rxode2 2.0.11 using 4 threads (see ?getRxThreads)mod1 <- rxode2({
C2 <- centr/V2;
C3 <- peri/V3;
d/dt(depot) <- -KA*depot;
d/dt(centr) <- KA*depot - CL*C2 - Q*C2 + Q*C3;
d/dt(peri) <- Q*C2 - Q*C3;
d/dt(eff) <- Kin - Kout*(1-C2/(EC50+C2))*eff;
})5.1指定ODE参数和初始条件
模型参数可以定义为命名向量,向量中参数的名称必须是ODE模型中参数的超集【译者注:即ODE中的参数是向量中参数名称的子集】,向量内参数的顺序并不重要。
theta <-
c(KA=2.94E-01, CL=1.86E+01, V2=4.02E+01, # central
Q=1.05E+01, V3=2.97E+02, # peripheral
Kin=1, Kout=1, EC50=200) # effects初始条件(ICs,Initial conditions)也可以通过向量定义。如果未指定元素,则假定房室的初始条件为零。
inits <- c(eff=1)如果要在模型中指定初始条件,可以添加:
eff(0) = 15.2在rxode2中指定给药和采样
rxode2提供了一种简单且非常灵活的方式,通过生成事件表的函数来指定给药(Dosing)和采样(Sampling) 。首先,通过”eventTable()“函数生成一个空的事件表:
ev <- eventTable(amount.units='mg', time.units='hours')接下来,使用add.dosing()和add.sampling()函数 EventTable对象来指定给药(数量、频率和/或时间等)和对系统状态进行采样观测的时间。这些函数可以多次调用以指定更复杂的给药或取样方案。在这里,使用下述代码通过这些函数指定10mg BID给药5天,随后20mg QD给药 5天:
ev$add.dosing(dose=10000, nbr.doses=10, dosing.interval=12)
ev$add.dosing(dose=20000, nbr.doses=5, start.time=120,
dosing.interval=24)
ev$add.sampling(0:240)如果您愿意,您也可以使用mattigr管道操作符%>%来执行此操作
ev <- eventTable(amount.units="mg", time.units="hours") %>%
add.dosing(dose=10000, nbr.doses=10, dosing.interval=12) %>%
add.dosing(dose=20000, nbr.doses=5, start.time=120,
dosing.interval=24) %>%
add.sampling(0:240)函数get.dosing()和get.sampling()可用于从事件表中检索信息。
head(ev$get.dosing())#> id low time high cmt amt rate ii addl evid ss dur
#> 1 1 NA 0 NA (default) 10000 0 12 9 1 0 0
#> 2 1 NA 120 NA (default) 20000 0 24 4 1 0 0head(ev$get.sampling())#> id low time high cmt amt rate ii addl evid ss dur
#> 1 1 NA 0 NA (obs) NA NA NA NA 0 NA NA
#> 2 1 NA 1 NA (obs) NA NA NA NA 0 NA NA
#> 3 1 NA 2 NA (obs) NA NA NA NA 0 NA NA
#> 4 1 NA 3 NA (obs) NA NA NA NA 0 NA NA
#> 5 1 NA 4 NA (obs) NA NA NA NA 0 NA NA
#> 6 1 NA 5 NA (obs) NA NA NA NA 0 NA NA您可能会注意到,这些与NONMEM事件表类似;如果您更熟悉NONMEM数据和事件,您可以直接将它们与事件表函数et一起使用
ev <- et(amountUnits="mg", timeUnits="hours") %>%
et(amt=10000, addl=9,ii=12,cmt="depot") %>%
et(time=120, amt=2000, addl=4, ii=14, cmt="depot") %>%
et(0:240) # Add sampling 您可以从上面的代码中看到,您可以将给药分配到rxode2模型中已命名的房室。这种与NONMEM的轻微偏差可以减少房室重新编号的需要。
这些事件也可以通过rbind、c、seq和rep组合和扩展(到多个体事件和复杂的方案)。有关使用rxode2创建复杂给药方案的更多信息,请参阅rxode2事件部分。
5.3求解ODE
现在可以通过调用模型对象的run或solve函数求解。将模型中所有变量的模拟结果存储在输出矩阵x中。
x <- mod1$solve(theta, ev, inits);
knitr::kable(head(x))| time | C2 | C3 | depot | centr | peri | eff |
|---|---|---|---|---|---|---|
| 0 | 0.00000 | 0.0000000 | 10000.000 | 0.000 | 0.0000 | 1.000000 |
| 1 | 44.37555 | 0.9198298 | 7452.765 | 1783.897 | 273.1895 | 1.084664 |
| 2 | 54.88296 | 2.6729825 | 5554.370 | 2206.295 | 793.8758 | 1.180825 |
| 3 | 51.90343 | 4.4564927 | 4139.542 | 2086.518 | 1323.5783 | 1.228914 |
| 4 | 44.49738 | 5.9807076 | 3085.103 | 1788.795 | 1776.2702 | 1.234610 |
| 5 | 36.48434 | 7.1774981 | 2299.255 | 1466.670 | 2131.7169 | 1.214742 |
您也可以求解这模型并创建一个rxode2数据框:
x <- mod1 %>% rxSolve(theta, ev, inits);
x#> ── Solved rxode2 object ──
#> ── Parameters (x$params): ──
#> V2 V3 KA CL Q Kin Kout EC50
#> 40.200 297.000 0.294 18.600 10.500 1.000 1.000 200.000
#> ── Initial Conditions (x$inits): ──
#> depot centr peri eff
#> 0 0 0 1
#> ── First part of data (object): ──
#> # A tibble: 241 × 7
#> time C2 C3 depot centr peri eff
#> [h] <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
#> 1 0 0 0 10000 0 0 1
#> 2 1 44.4 0.920 7453. 1784. 273. 1.08
#> 3 2 54.9 2.67 5554. 2206. 794. 1.18
#> 4 3 51.9 4.46 4140. 2087. 1324. 1.23
#> 5 4 44.5 5.98 3085. 1789. 1776. 1.23
#> 6 5 36.5 7.18 2299. 1467. 2132. 1.21
#> # … with 235 more rows这将返回一个修改后的数据框。您可以在下图中看到房室中的数值:
library(ggplot2)
plot(x,C2) + ylab("Central Concentration")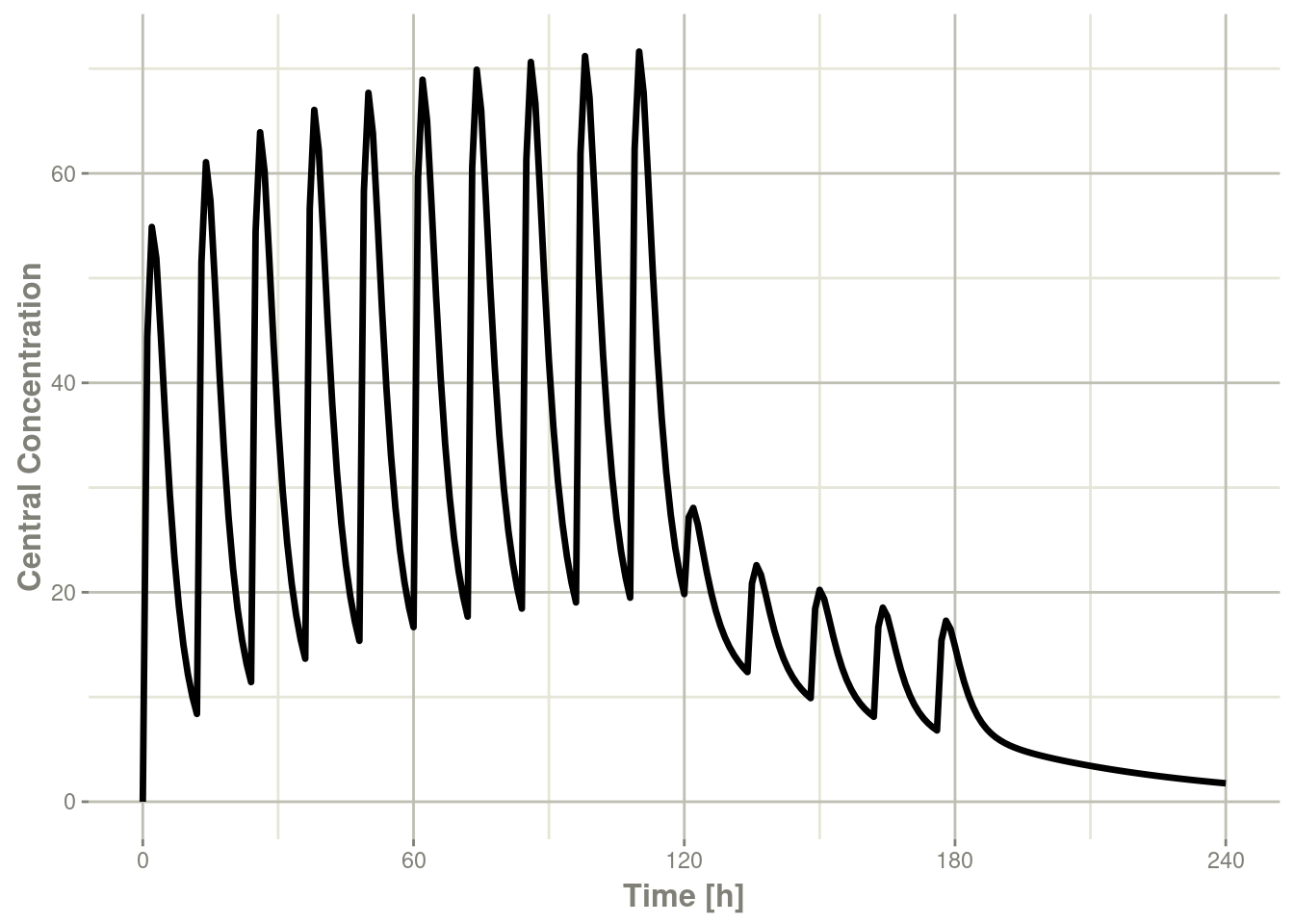
或者,
plot(x,eff) + ylab("Effect")
请注意,标签会自动使用初始事件表中的单位进行标记。rxode2提取units来标记绘图(如果它们存在)。